DNAも言語だ!生成AIの言語モデルとバイオテクノロジーの関係

皆さんは、ChatGPTやGitHub Copilotなどの生成AI技術を活用していますか?
高度な質の応答や、コーディング補助で便利に活用されている生成AI技術は、実は生命科学という一見関係のない分野においても現在非常に注目を集めています。今回は、なぜ生成AI技術が生命科学において注目されているのか?最新の研究事例は何か?などを、最新の研究事例を交えながら解説します。
筆者は、普段ライフサイエンスやコンピュータサイエンスを専門とするバイオインフォマティシャンで、主にDNAなどの情報解析をしている博士学生です。この記事では生命科学と、ChatGPTをはじめとする大規模言語モデルの意外な関係性を、普段の研究分野や最新の事例を交えながら解説します。関連する記事も、ぜひご覧ください。
大規模言語モデルがやっているのは「穴埋め問題」
まず、大規模言語モデル(LLM)とは何かを見てみましょう。大規模言語モデルは何をしているかというと、「単語の補完問題」を解いています。イメージとしては以下の図のように、文章が与えられたときに、一部については穴埋めしている状況で、そこの穴に何が入るかを予測しています。
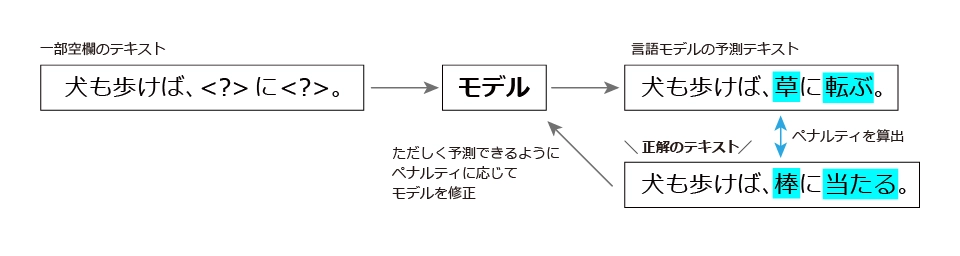
図のように、モデルが誤った予測を出すことに対してペナルティがつき、このペナルティを減らすようにモデル関数のパラメータを更新していきます。これにより、コンピューターがゼロからテキスト表現を学習していきます。
この作業のことを事前学習といい、一般に大量のデータ数と大量のGPUを必要とする高負荷の計算が処理されます。モデルの学習のさらなるイメージについては、以下の記事で解説しているので、こちらもぜひ読んでみてください。
現在、事前学習済みモデルはオープンソース化されて、誰でも簡単にアクセスして使えるようになっています。Hugging Faceという公開型のAIコミュニティーでは、開発者が訓練に使用したデータセットやモデル自体をオープンソースにアップロードしているので、誰でも利用できます。
▼Hugging Face – The AI community building the future.
https://huggingface.co/
生命科学における「言語」— それはDNA
大規模言語モデルが活躍する舞台は、何も私たちが一般に読み書きする自然言語だけではありません。
そもそも「言語」という時に、どのような定義ができるでしょうか?言語は記号からできていることを、ぱっと思いついた人もいるでしょう。言葉は「あいう」といった文字記号で表現されますね。言語は、情報伝達手段です。情報を伝達し、共有するためのシステムですね。そして、文法があります。構文や文法規則に従って、記号を組み合わせることで意味のある表現を作り出します。どんな例でも、例えばそれがとても少数話者の言語だろうが、超マイナーなプログラミング言語だろうが、言語と言えるものは、上記のような条件を満たしているのではないでしょうか。
そして、生命の遺伝情報を司るDNAも、以下の観点から、非常に言語に近い性質を持っていることが分かります。
記号:DNAは4つの記号から構成される
DNAとは、4つの塩基(アデニン[A]、チミン[T]、グアニン[G]、シトシン[C]の4種類のヌクレオチド)の配列のことです。
情報伝達:DNAは生命の遺伝情報を司る
DNAは『生命の設計図』と呼ばれ、セントラルドグマという原理に基づき、その遺伝情報は親から子へ引き継がれます。DNAとは、情報そのものです。
文法:制御文法がある
DNAから、遺伝情報を発現したんぱく質が産生されますが、これはDNAの持つ高度な文法規則に支配されています。プロモーターやエンハンサーといったDNAの特定の領域は、遺伝子の発現を開始するための「スタートコード」として機能し、スプライシングといったプロセスは、RNAメッセージの「編集」に相当します。
これは言語が持つ文法に類似していて、例えば文字の並びによって文章の意味が変わるように、DNAも塩基の並びによって生物学的な意味が変わってきます。
大規模言語モデルによるDNA言語の学習
ここまで来ると、DNAもやはり、大規模言語モデルでの学習対象になることが予想できます。実際、DNA言語モデルの開発は最先端の研究分野です。大規模言語モデルの考え方を用いて、実際に以下のようなイメージでDNA言語が学習されます。
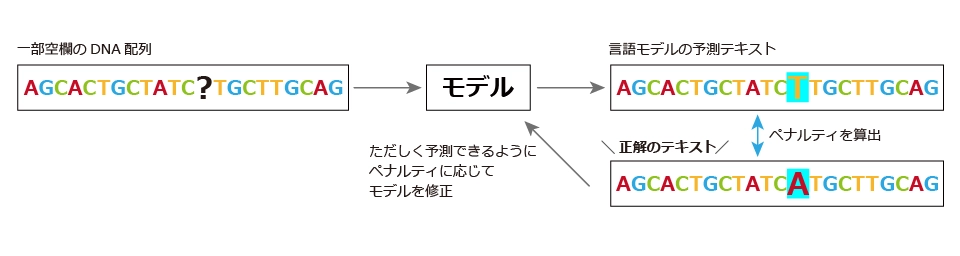
このように、DNA言語は、自然言語と同じような枠組みで学習されます。ヒト全ゲノムに関しては、塩基文字が全体で約30億塩基対あると言われています。そのため、30億塩基対全体の情報をカバーするように計算すると、そのゲノムコンテキスト全体を学習できます。
DNA言語モデルの研究事例
DNABERT
DNABERTは、自然言語処理(NLP)で広く用いられるBERT(Bidirectional Encoder Representations from Transformers)を、DNA配列データに適用したモデルです。BERTモデルは、文脈に依存する単語の埋め込みを生成することで、言語の深い理解を可能にします。
DNABERTでは、このアプローチをDNA配列に適用し、DNAの各塩基(アデニン[A]、チミン[T]、グアニン[G]、シトシン[C])や塩基配列を「単語」と見なして扱います。一部の塩基をマスクして隠し、モデルがそのマスクされた塩基を予測するように事前学習します。現在は、2021年版から改良されたバージョンも出ています。
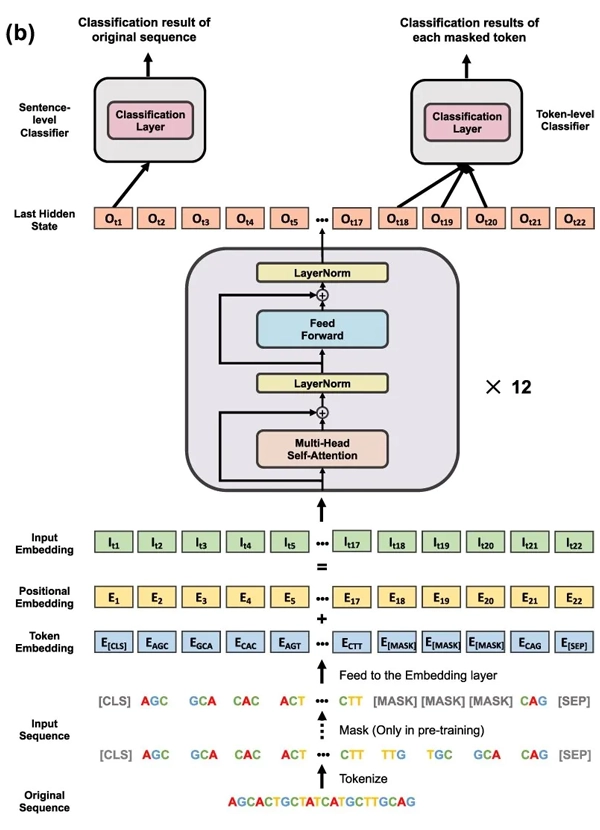
出典:Ji, Yanrong, et al. “DNABERT: pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome.” Bioinformatics 37.15 (2021): 2112-2120.
Enformer
Enformerは、DeepMindによって開発された、遺伝子発現の予測に特化したモデルです。このモデルは、言語表現を事前学習するわけではなく、DNA配列の長い範囲を入力として取り込み、特定の領域での遺伝子発現量を学習します。
Enformerは、Transformerベースのアーキテクチャを使用しており、DNA配列の大域的な文脈と局所的な文脈の両方を捉えられます。
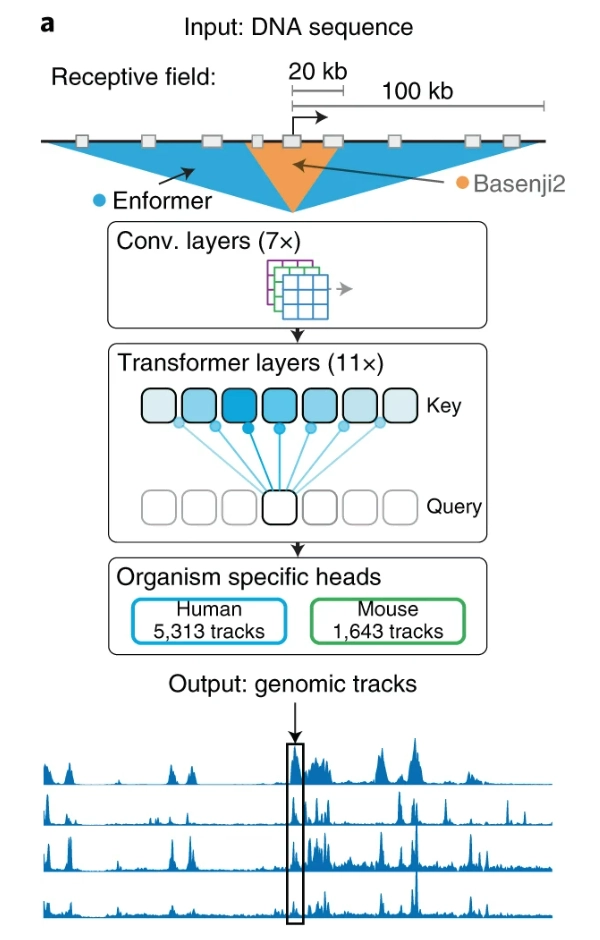
出典:Avsec, Žiga, et al. “Effective gene expression prediction from sequence by integrating long-range interactions.” Nature methods 18.10 (2021): 1196-1203.
最新の研究トレンドは、より長い塩基配列の学習
現在、多くのTransformerベースモデルがDNA言語学習用に開発されています。
DNAの場合、事前学習の計算コストを決めるのは配列長(一度に処理する配列の長さ)です。しかし、現在のTransformerをベースとしたモデルはアルゴリズムの性質上、長い配列にした時(例えば100万bp)に掛かるGPU負荷が原因で、学習効率が悪くなることが知られています。
そのため、Transformerベースのアーキテクチャから脱却して、HyenaDNAなどのより計算効率の高いアーキテクチャ設計に移行することで、さらに長い配列長を処理する流れになりつつあります。非常に技術進展の速い分野なので、キャッチアップが必要ですね。
2024年2月現在の最新のDNA言語モデル(HyenaDNA, Evo)はすでに、10 万 – 100万塩基長を、一度に処理できるところまできています。2021年のDNABERTモデルでは、せいぜい500塩基長ぐらいまでしか処理できなかったのに比較すると、3年で1000倍以上の長さの配列を処理できるようになっています。
この技術的革新を支えているのは、モデル内部で使われている行列計算アルゴリズムの進化です。従来のTransformerは今後使われなくなるのではないか、と噂になっています。
生命科学解析と大規模言語モデルはさらに密接に
今回は、生命科学における大規模言語モデルについて、実際の研究事例を交えて紹介しました。DNAに限らず、生命科学は言語の宝庫(例えばAlphaFoldで有名なアミノ酸配列など)です。今後ますます、生命科学解析と大規模言語モデルが密接に関係していくことは明らかでしょう。
参考文献
▼Yanrong Ji, Zhihan Zhou, Han Liu, Ramana V Davuluri, DNABERT: pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome, *Bioinformatics*, Volume 37, Issue 15, August 2021, Pages 2112–2120
https://academic.oup.com/bioinformatics/article/37/15/2112/6128680
▼Nguyen, Eric, et al. “Hyenadna: Long-range genomic sequence modeling at single nucleotide resolution.” *Advances in Neural Information Processing Systems* 36 (2024).
https://proceedings.neurips.cc/paper_files/paper/2023/hash/86ab6927ee4ae9bde4247793c46797c7-Abstract-Conference.html
▼Evo: Long-context modeling from molecular to genome scale
Evo: Long-context modeling from molecular to genome scale (together.ai)











四葉さん、コメントいただきありがとうございます。にんじんです。 僕がこの会社この…
面白い話をありがとうございます。私自身は法学部ですが哲学にも興味があります。 ふ…
要所要所に別記事が挿入されていて読みやすかったです! 文系の人でもバイオインフォ…
お読みいただきありがとうございました。最果タヒさんの作品は、詩集だけでなくエッセ…
萩原朔太郎や宮沢賢治の詩集が好きなのですが、同じ読書という行為であっても小説を読…